非線形最小二乗法で学習コンテンツ消化曲線をモデル化
まあ結構いろんなことに汎用的に当てはまることだと思うのだけど,ソフトウェアの品質管理とかの分野では,ソフトウェア信頼度成長曲線という手法があるそうだ(古い友人に教えてもらった)。
これは,ソフトウェア開発において,バグの発見数や残ったバグの数の予測などに使われるものだそうで,基本的には横軸に時間や工数,縦軸にはバグの数とかをプロットする。すると,S字カーブのような曲線になるそうだ。で,これを非線形最小二乗法を使って何かの関数に当てはめてモデル化するというはなし。ふむふむ。とっても面白そう。品質管理というだけあって,実用的だし,外国語教育の業務改善にもすごく通じるものがありそうだ。
外国語教育では,オンライン教材におけるコンテンツ消化率とかがパッと思いつくような例だ。オンライン教材で単語テストを繰り返すとか,そういう場面では,横軸に時間を,縦軸に消化率をプロットすると,おそらく同じように成長曲線を描くことができる。こんな感じだということしよう。ある学生があるオンライン教材を15週にかけて勉強した様子で,横軸には週,縦軸にはそのコンテンツの消化率をプロットした,と。(こういうデータは外国語教育にはかなりお蔵入りしてる)
#例の作成 t<-seq(1,15,1) d<-c(0.04,0.02,0.03,0.06,0.12,0.22,0.36,0.50,0.60,0.72,0.85,0.91,0.97,0.98,1.00) #作図 plot(t,d,xlab="時間(単位:週)",ylab="コンテンツ消化率")

このデータになんらかの関数を当てはめたい,と(最近欲求不満なのかなんでも当てはめたくなる)。
えっと,いろんな関数が当てはまるだろうけど,ここでは,ゴンペルツと4母数ロジスティック関数にしよう。
Rでやるには,デフォルトで使えるstatsパッケージのnlsという関数を使えばいい。非線形最小二乗法でフィッティングをする関数だ。いろいろ便利。
#ゴンペルツに当てはめ fit.gom<-nls(d~SSgompertz(t,Asym,b2,b3)) #結果 summary(fit.gom) Nonlinear regression model model: d ~ SSgompertz(t, Asym, b2, b3) data: parent.frame() Asym b2 b3 1.0985 12.1508 0.7114 residual sum-of-squares: 0.004886 Number of iterations to convergence: 0 Achieved convergence tolerance: 5.022e-06 AIC(fit.gom) BIC(fit.gom) #当てはめた曲線を描き足し plot(t,d,xlab="時間(単位:週)",ylab="コンテンツ消化率") lines(t,predict(fit.gom),lty=2,col=2) #4母数ロジスティック関数に当てはめ fit.fpl<-nls(d~SSfpl(t,A,B,xmid,scal)) #結果 summary(fit.fpl) Formula: d ~ SSfpl(t, A, B, xmid, scal) Parameters: Estimate Std. Error t value Pr(>|t|) A -0.009647 0.015871 -0.608 0.556 B 1.023469 0.017314 59.114 4.01e-15 *** xmid 8.223108 0.108640 75.692 2.66e-16 *** scal 1.791406 0.112481 15.926 6.06e-09 *** --- Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 Residual standard error: 0.01943 on 11 degrees of freedom Number of iterations to convergence: 0 Achieved convergence tolerance: 3.818e-06 AIC(fit.fpl) BIC(fit.fpl) #当てはめた曲線をさらに描き足し plot(t,d,xlab="時間(単位:週)",ylab="コンテンツ消化率") lines(t,predict(fit.gom),lty=2,col=2) lines(t,predict(fit.fpl),lty=2,col=4) #2つのモデルをANOVA anova(fit.gom,fit.fpl) Analysis of Variance Table Model 1: d ~ SSgompertz(t, Asym, b2, b3) Model 2: d ~ SSfpl(t, A, B, xmid, scal) Res.Df Res.Sum Sq Df Sum Sq F value Pr(>F) 1 12 0.0048859 2 11 0.0041547 1 0.00073119 1.9359 0.1916 #予測のために #ゴンペルツ曲線の関数を定義 gomf<-function(x,Asym,b2,b3){ y<-Asym*exp(-b2*b3^x) y } #4母数ロジスティック曲線の関数を定義 fplf<-function(x,A,B,xmid,scal){ y<-A+(B-A)/(1+exp((xmid-x)/scal)) y }
この例だとピッタシだ。

外国語教育研究では,これと似たような技術の応用に,キーログを使った川口ほか(2016, Language Education & Technology)がある。
CiNii 論文 - エッセイライティングにおける増加語数の時系列推移傾向とエッセイ評価の関係 : モデルフィッティングを用いた検討
混合正規指数合成分布モデル(?)を最尤法で…
聞いたこともないけど,要素数2の混合正規指数合成分布(ex-Gaussian)モデルというのを考えてみる。ま,2つの異なる認知プロセスが混合したときの反応時間の分布だとか,そんなそれっぽいことを考えてみる。そんなものは多分ない。
ま,でもこの確率密度関数は,
dmixexgauss<-function(x,lambda,mu1,sigma1,tau1,mu2,sigma2,tau2){ y<-lambda*(1/tau1)*exp(mu1/tau1+(sigma1^2)/(2*tau1^2)-x/tau1)* pnorm(x,mu1+(1/tau1)*sigma1^2, sigma1) +(1-lambda)*(1/tau2)*exp(mu2/tau2+(sigma2^2)/(2*tau2^2)-x/tau2)* pnorm(x,mu2+(1/tau2)*sigma2^2,sigma2) return(y) }
こうなはず。これで尤度関数を作って,bbmleパッケージのmle2とかで最尤推定してみれば…と思ったんだけど,やっぱ上手く推定できなかった。methodもBFGSとかいろいろ試したけど,ううむ。なんと7母数の確率密度関数だもんな。俺の間違いってのもあるし。
ま,でもこの自作の確率密度関数自体はあってるぽい。暇ができたらちょっとやってみよう。
library(retimes) set.seed(1) dat<-c(rexgauss(400,1000,300,800),rexgauss(100,4000,800,500)) hist(dat,main="",breaks=20,col="lightblue",freq=F,ylim=c(0,.0006),xlim=c(0,8000)) x<-seq(0,8000,1) lines(x,dmixexgauss(x,.80,1000,300,800,4000,800,500),lty=2,col=2)
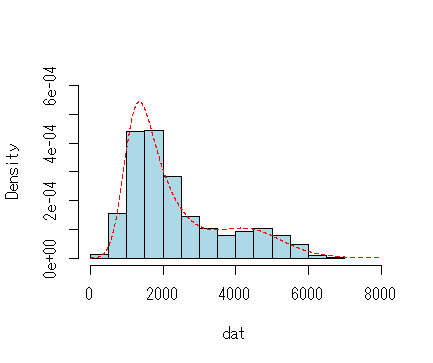
*追記(1/28)
標本サイズ,母数,初期値次第では結構うまくいく場合もあるようだ。たとえば,
#あからさまな例 set.seed(1) dat<-c(rexgauss(500,1000,500,100),rexgauss(500,10000,500,100)) #mle2で最尤推定 #対数尤度を返す関数 lf<-function(l,m1,s1,t1,m2,s2,t2){ -sum(log(dmixexgauss(dat,l,m1,s1,t1,m2,s2,t2))) } #適当な初期値で最尤推定 fit<-mle2(lf,start=list(l=.01,m1=1000,s1=100,t1=100,m2=10000,s2=100,t2=100)) summary(fit) Maximum likelihood estimation Call: mle2(minuslogl = lf, start = list(l = 0.01, m1 = 1000, s1 = 100, t1 = 100, m2 = 10000, s2 = 100, t2 = 100)) Coefficients: Estimate Std. Error z value Pr(z) l 5.0001e-01 1.5811e-02 31.6234 < 2.2e-16 *** m1 8.8352e+02 5.3678e+01 16.4598 < 2.2e-16 *** s1 4.2277e+02 2.8256e+01 14.9618 < 2.2e-16 *** t1 2.5258e+02 5.1428e+01 4.9114 9.043e-07 *** m2 9.8383e+03 5.6083e+01 175.4242 < 2.2e-16 *** s2 4.4467e+02 2.8829e+01 15.4245 < 2.2e-16 *** t2 2.4854e+02 5.3408e+01 4.6536 3.263e-06 *** --- Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1 -2 log L: 16646.24 coef(fit) vcov(fit) AIC(fit)
当てはめた分布と推定した母数から平均,分散,歪度,尖度をもとめる
なんかある論文で,分布が強く歪んでいることが理論的に明白だった変数に,ガンマ分布か対数正規分布かなにかを当てはめて,その推定母数と適合度指標のみを(きつい紙幅の関係もあって)報告したときに,「標本の記述統計を報告しないとはけしからん」というようなことを査読者さまにご教示されたことがある。ま,ごもっともであるけど,紙幅の関係もあって,ううむ,標本自体がそんな重要なのかな…みたいに思いつつも,どうしたんだったかな。結構前の話だ。
でも,もちろん標本の記述統計量はわからんけど,この当てはめた分布の母数の推定値から,理論的には,そしてその分布モデルがしっかりと当てはまっているなら,標本のおおよその記述統計であれば,あとからでもとめることもできる。たとえばガンマ分布はかなり簡単で,こんな自作関数でも作ればいい。
gammadescriptive<-function(shape,scale){ m<-shape*scale v<-shape*(scale^2) s<-2/sqrt(shape) k<-6/shape result<-list("mean"=m,"variance"=v,"skew"=s,"kurtosis"=k) result }
たとえば,この関数に,形状母数3,尺度母数(scale)4を入れてやる。
gammadescriptive(3,4) $mean [1] 12 $variance [1] 48 $skew [1] 1.154701 $kurtosis [1] 2
するとこんな感じになる。もちろん,「あくまでも理論値」なんだけど。
こんな感じで実験してみよう。
#パッケージの準備 library(psych) library(fitdistrplus) #例の作成 set.seed(1) dat<-rgamma(1000,shape=3,scale=4) #理論値 gammadescriptive(3,4) $mean [1] 12 $variance [1] 48 $skew [1] 1.154701 $kurtosis [1] 2 #この例の記述統計を見てみる describe(dat) vars n mean sd median trimmed mad min max range skew kurtosis se X1 1 1000 11.95 7.09 10.4 11.19 6.59 0.74 50.2 49.46 1.12 1.86 0.22 #大体近い #また,この乱数にガンマ分布をフィットさせる model<-fitdist(dat,"gamma") model Fitting of the distribution ' gamma ' by maximum likelihood Parameters: estimate Std. Error shape 2.8585896 0.12111673 rate 0.2392977 0.01108254 #ちっ,これrateで出してきやがる,でもscale=1/rate #でも,大体形状母数3,尺度母数4くらい sh<-model$estimate[[1]] sc<-1/model$estimate[[2]] #またこの母数の理論値を見てみる gammadescriptive(sh,sc) $mean [1] 11.94575 $variance [1] 49.92004 $skew [1] 1.182917 $kurtosis [1] 2.098937
もちろんだけど,おおよそ標本の記述統計にも合っている。
だから,ある変数に狙った分布がよく当てはまっているといえるのであれば,平均,分散,歪度,尖度などは報告しなくても,「後からおおよその値は計算できる」こともあることは事実。ただ,この記述統計4つを報告したところで,この経験分布はガンマ分布に近いとか適合度はどれくらいだとか,そういうのはわからないか,または技術的により困難だってこと。
もちろん,標本の記述統計は要りません,って話じゃないけど。
混合ガンマ分布モデルをデータにフィットさせる
Rのmixtoolsパッケージでは,混合ガンマ分布モデルもデータへフィットさせることができる。
#λ= .25,.75,α - 4, 10,β= 1, .2が正解 set.seed(0) dat<-c(rgamma(100,4,1),rgamma(300,10,.2)) hist(dat,breaks=20, main="",col="lightblue") model<-gammamixEM(dat) model$gamma.pars model$lambda
まあまあ,て感じ。これ結構混ざっちゃうと,うまくいかないかもね。
混合ガンマ分布の確率密度関数は,
dmixgamma<-function(x,lambda,a1,a2,b1,b2){ y<-lambda*dgamma(x,a1,b1)+(1-lambda)*dgamma(x,a2,b2) y }
これで絵を描くとこう。
hist(dat,breaks=20, main="",col="lightblue",freq=F,ylim=c(0,.06),xlim=c(0,100)) x<-seq(0,100,.01) lines(x,dmixgamma(x,0.25,3.05,11.54,1/1.23,1/4.37),lwd=2,col=2)

…これ,単変量で,しかも前もって2つの混合だってわかっているんだったら,EMアルゴリズムでなくても,この程度の単純さなら普通の(?)L-BFGSとかでいけそうだよね。
そしたら結局はfitdist,mle,bbmle,optimとかでも同じことできそう。
指数正規合成分布とガンマ分布の混合とか,そういうのも自由にできるはず。おもったより簡単ぽい。
単変量混合正規分布モデルをデータにフィットさせる
外国語教育研究では2つの山とか3つの山とかの分布になっているのを見ることがある。こういうときは,混合分布モデルをデータにフィットさせるといいかもだ。Rではmclustもいいけど,mixtoolsというパッケージがある。
#準備 library(mixtools) #数値例の作成 #ここでは混合比(λ)が1:2,母平均(μ)が50, 120,母標準偏差(σ)が10, 20が正解 set.seed(92) huta<-c(rnorm(100,50,10),rnorm(200,120,20)) #可視化して確認 par(mfrow=c(1,2)) hist(huta,col="lightgray",xlab="Value",main="") plot(ecdf(huta))

#mixtoolsのEMアルゴリズム関数を使って母数を推定 fit<-normalmixEM(huta) summary(fit) summary of normalmixEM object: comp 1 comp 2 lambda 0.669647 0.330353 mu 117.657305 50.067067 sigma 20.090447 9.126634 loglik at estimate: -1428.916 #ここではだいたい正解だった #対数尤度 fit$loglik #AIC (自由母数は5個でいいよね?) fitaic<--2*fit$loglik+2*5 fitaic #誤差をブートストラップで boot.se(fit) #ヒストグラムに確率密度関数を合わせる hist(huta,col="lightgray",xlab="Value",main="",freq=F,ylim=c(0,.015),breaks=20) #その前に推定した母数をもつ混合正規分布の確率密度関数を定義する dnormmix<-function(x){ y<-fit$lambda[1]*dnorm(x,fit$mu[1],fit$sigma[1])+fit$lambda[2]*dnorm(x,fit$mu[2],fit$sigma[2]) y } x<-seq(0,200,0.1) lines(x,dnormmix(x),lwd=2,col=2)

うむ。間便な混合正規分布の確率密度関数も書いておく。これで好きにお絵かきできる。
dnormmix<-function(x,lambda, mu1, mu2, sigma1, sigma2){ y<-lambda*dnorm(x,mu1,sigma1)+(1-lambda)*dnorm(x,mu2,sigma2) y }
(なぜか混合分布モデルは自分の青春だった,って感じがする)
任意の累積分布関数を仮定した一標本コルモゴロフ・スミルノフ検定
#正規分布の場合 set.seed(0) dat<-rnorm(100,0,1) ks.test(dat,"pnorm") #特定の平均と標準偏差をもつ正規分布 set.seed(0) dat<-rnorm(100,0,1) ks.test(dat,"pnorm",1,1) #ガンマ分布 set.seed(0) dat<-rgamma(100,2,3) ks.test(dat,"pgamma",2,3) #ワイブル分布 set.seed(0) dat<-rweibull(100,2,3) ks.test(dat,"pweibull",2,3)
コルモゴロフ・スミルノフ検定で大事なのは,(よく正規性の検定に使われるけど)帰無仮説は任意の累積分布関数であって,その帰無仮説は自由に立てられる(一般に正規性の検定で使われる方法は,標本から推定した母平均と母標準偏差を項にもつ正規分布の累積分布関数を帰無仮説として設定している,ということ)。帰無仮説を棄却できないとき,「標本がその累積分布関数に適合していないとはいえない」だけであって,積極的に任意の累積分布関数から発生した標本だとか,母集団の分布はまさにこれだとか,ちょっと厳しくいえば正規分布に従っている,とか,そういうように語気強くいうべきではないかも。もちろん,正規性を示すひとつの証拠であることは間違いないけど。
自分で用意した分布をデータへフィットさせる
fitdistrplusパッケージのfitdist関数は,dnorm, pnormのように,dとpの関数が定義されていればどんなものでも指定することができる。自作でもいいってこと。ま,でも殆どの場合,自分程度が思いつくような分布はすでに用意されているってのがR。
たとえば,レイリー分布の場合。レイリー分布はVGAMパッケージに含まれている。レイリー分布のパラミタはひとつ(形状母数)。
#パッケージの読み込み library(VGAM) #例の作成 dat<-rrayleigh(1000,300) #fitdist関数によるフィット #初期値を指定してやらなければならない。とりあえずここではデータの中央値を入れてやる。 fit<-fitdist(dat,"rayleigh",start=list(scale=median(dat))) summary(fit) Fitting of the distribution ' rayleigh ' by maximum likelihood Parameters : estimate Std. Error scale 296.7956 4.692907 Loglikelihood: -6652.58 AIC: 13307.16 BIC: 13312.07 plot(fit)

よし。
じゃ,次は指数正規合成分布。これはretimesパッケージにある。ただし,dexgaussとrexgaussしかないので,pexgaussを自分で作ってしまう。
#パッケージ読み込み library(retimes) #pexgaussを作成 pexgauss<-function(x,mu,sigma,tau){ tau<-1/tau u<-tau*(x-mu) v<-tau*sigma phi1<-pnorm(u,0,v) phi2<-pnorm(u,v^2,v) e<-exp(1) p<-phi1-e^(-u+(v^2)/2+log(phi2)) p } #数値例の作成 dat<-rexgauss(1000,1000,500,1000) #fitdist関数によるフィット #初期値を指定してやらなければならない。とりあえずここではデータの正解を入れてやる。 fit<-fitdist(dat,"exgauss",start=list(mu=1000,sigma=500,tau=1000)) summary(fit)
ちなみに,retimesパッケージにはtimefitっていう専用の関数がある。ま,ほぼおなじ推定値が帰ってくる。
timefit(dat)
なんかもうoptimやりゃいいって気がしてくるな。